近日,计算机与信息学院李召军博士团队联合中国科学院上海药物研究所和上海科技大学在《核酸研究》(Nucleic Acids Research,影响因子14.9)上发表了题为“KinomeMETA: a web platform for kinome-wide polypharmacology profiling with meta-learning”的文章,构建了一个支持用户定制的激酶多靶点活性筛选AI平台KinomeMETA(https://kinomemeta.alphama.com.cn/,图1)。该平台使用结合图神经网络的元学习技术,极大地拓展了可准确预测的激酶范围,并为用户提供了使用私有数据定制模型的实用功能。
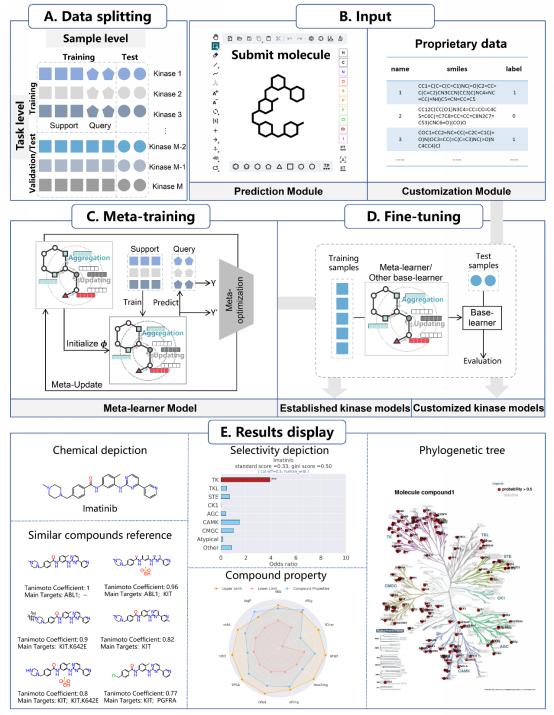
图1.KinomeMETA平台概览。(A)数据划分策略。(B)模块输入格式。其中预测模块输入分子结构。定制模块输入分子结构及其在目标激酶上的活性标签。(C)KinomeMETA的元训练过程。(D)KinomeMETA的微调过程。已建立的激酶模型通过微调元学习器得到。用户提交的私有数据根据研究目的用于微调元学习器或已建立的激酶模型。(E)预测模块的结果展示,包括相似结构的激酶抑制剂及其主要靶点,激酶选择性评估、系统发生树和分子性质。
KinomeMETA的预测(Prediction)模块可以预测用户提交分子对661个野生和突变激酶的抑制活性概率。与研究团队先前的小分子激酶多重药理学预测平台KinomeX(https://kinome.simm.ac.cn/en/)相比,KinomeMETA的训练数据和激酶预测范围得到了显著扩展——生物活性数据点增加了3.6倍,化合物增加了5倍,激酶增加了1.69倍。激酶可预测范围的增加得益于元学习算法,该算法使得KinomeMETA能够高效地利用稀疏活性数据对研究较少的激酶和突变激酶提供准确预测。KinomeMETA的用户定制(Customization)模块是平台的全新功能,该功能允许用户使用少量私有激酶活性数据在线建立定制模型(图1B,D)。这使得KinomeMETA摆脱了传统模型中仅能依靠公开实验数据构建的限制,帮助用户充分结合计算模型与已有实验数据,提升针对特定激酶的研究效能。
KinomeMETA是一个全面升级的激酶多靶点小分子抑制剂预测平台。相比于先前的KinomeX平台,KinomeMETA拥有更广泛的激酶预测范围和更高的预测精度。本平台的一个重要突破是引入了元学习驱动的定制模块。该模块使研究人员能够使用少量私有数据在线创建新激酶模型或改进现有激酶模型,通过迭代的干/湿实验循环,有效应对药物发现中的低数据场景。KinomeMETA平台将成为激酶研究人员的强力工具,显著加速新型激酶抑制剂的发现。
计算机与信息学院李召军博士、中国科学院上海药物研究所研究生曲宁和上海科技大学研究生周敬怡为论文的共同第一作者,德州学院为该成果的第1完成单位。中国科学院上海药物研究所李叙潼副研究员、郑明月研究员与张素林副研究员为论文通讯作者。
李召军博士团队长期致力于人工智能辅助药物设计的研究,在云计算、虚拟化技术与药物设计的结合领域取得了一系列成果,近几年在Nature Computational Science、Nucleic Acids Research、Bioinformatics、Journal of Medicinal Chemistry等国际知名期刊上发表研究成果近10篇。
(作者:李召军;供稿审核人:郭长友、王荣燕)
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkae380/7675173
KinomeMETA平台主页:
https://kinomemeta.alphama.com.cn

